我们用Gooey制作了一个蛋白配体分割工具。用以切割Pymol中保存的Autodock Vina对接结果。
工具下载地址: https://pan.baidu.com/s/1jIzGh2A 密码: kwg2
我们用Pymol插件中的Autodock Vina对接,对接完后在Pymol中将蛋白配体结果全部保存,保存以后问题来了,我们如何进行分析和后续处理呢?
比较简单的方法为进行手动切割,如图:
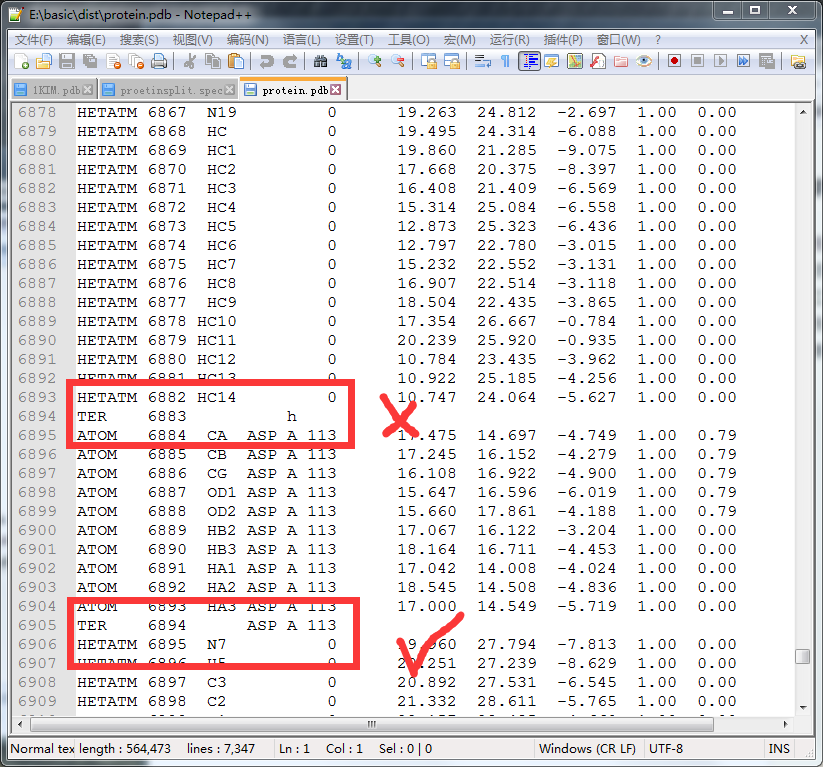
蛋白pdb中每一个配体均用TER\s+\d{3}\s+\W{3}\s的方式进行切割,所以我们仅需把最前面的蛋白提取,然后依次提取一段以TER分割的配体进去,比较麻烦。
另外我们不仅仅只使用Autodock Vina进行对接,可能还用到iGEMDOK(将在后期介绍)等对接软件进行对接。但是Pymol保存的Autodock Vina对接的结果中配体并无标识,这不利于后续分析。同时并没有HET蛋白配体标识,导致iGEMDOCK在内的对接软件无法定位。
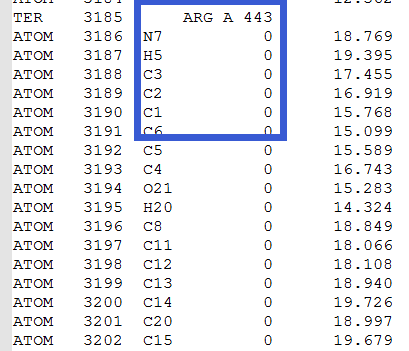
比较基础的方法既手动修改,比如第一行ATOM 3186 N7 0
修改为:ATOM 3186 N7 LIG D 999
,稍微高级一点可以使用正则进行匹配。这里不做详细介绍了。
最近采用Python+Gooey快速制作了一个蛋白配体分割工具。界面如图:
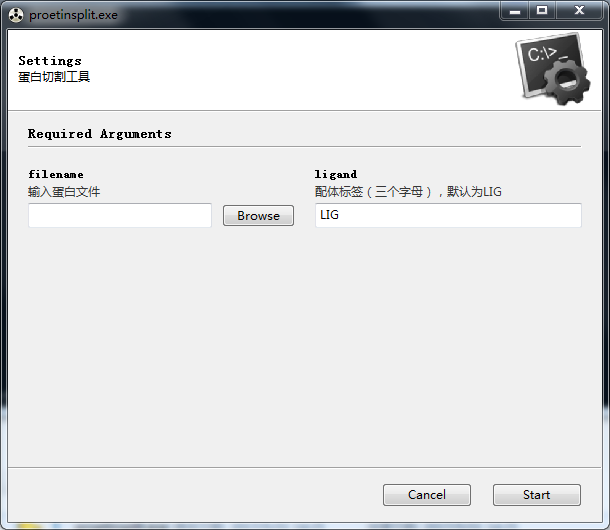
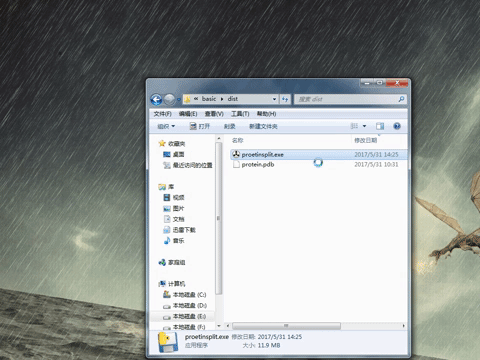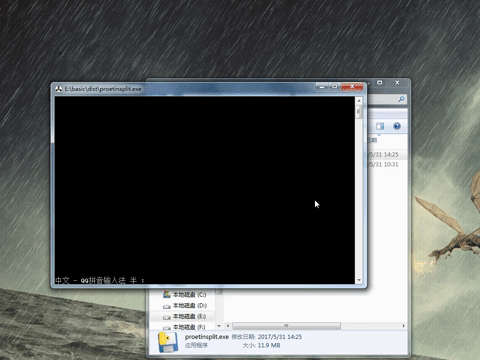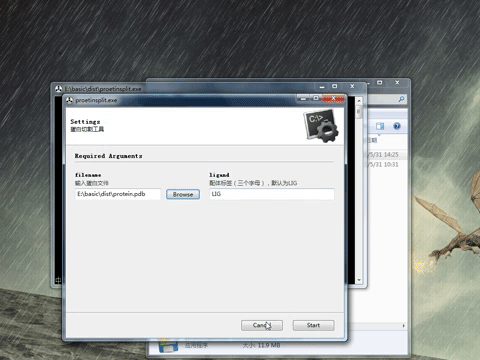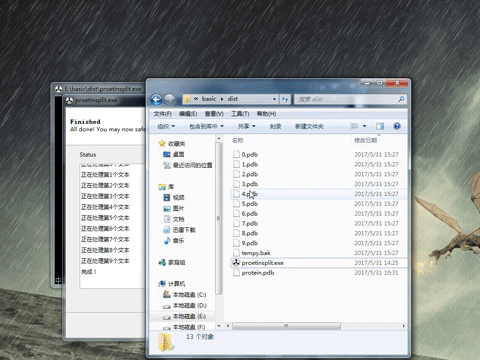
使用方法非常简单简便,用管理员身份打开软件。filename输入需要切割的pdb文件,右侧标签输入配体需要标记的标签,默认为LIG,最好使用三字母以免出错。
然后start,最后结果如下图:
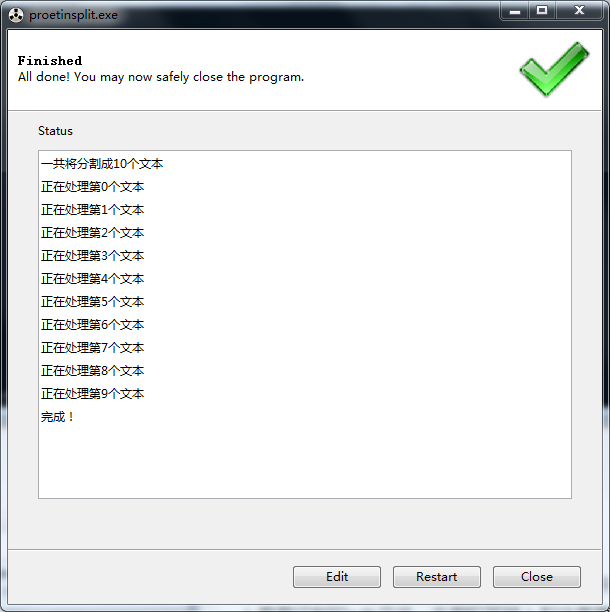
结果会以数字.pdb编号在软件运行目录下。
以下为完整教程:
非常简单方便
