杂志:Am J Cancer Res
影响因子:3.98
接收:2015年8月 发表:2015年11月
一篇伊朗的研究论文
题目:Molecular dynamics simulation on the low sensitivity of
mutants of NEDD-8 activating enzyme for
MLN4924 inhibitor as a cancer drug
全文下载:点我下载
摘要:MLN4924是NEDD8活化酶(NAE)抑制剂,而NAE是结合在culin蛋白上的库林环连接酶(CRLs)。如图:
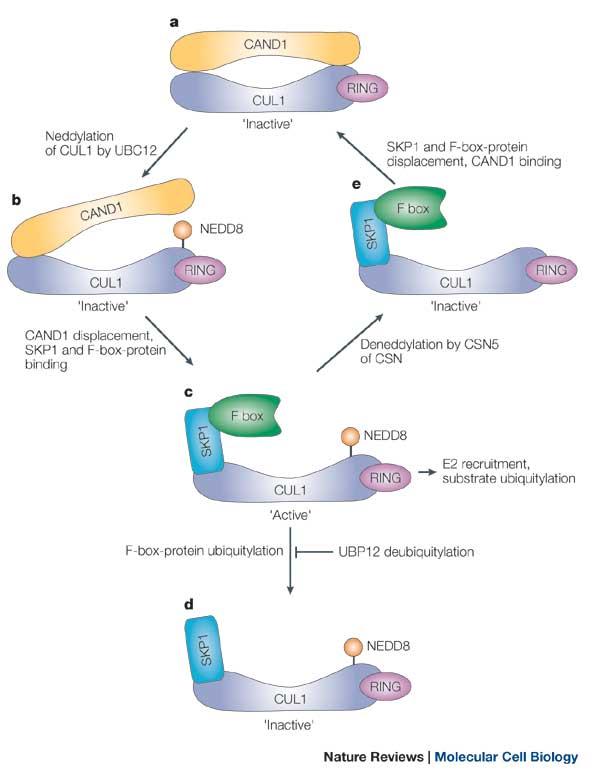
这个共价修饰招募E2泛素化调控原件,导致下游蛋白泛素化以及蛋白酶的降解。MLN4924为共价抑制剂!使得CLRs失活(而CLRs会导致DNA的复制以及抑制NF-κB信号通路)从而导致癌细胞的死亡。
突变可以减少酶对MLN4924的敏感性:Verma et al等人在2013年分子动力学研究发现A171T减少MLN4924和DNA结合位点作用酶( DNA Binding site of enzyme as a result of reduction
of enzyme affinity to ATP)的相互作用,减少酶和ATP的亲和力。
一年后Wei Xu et al.发现Y352H和I310N的突变导致酶阻碍MLN4924的抑制作用。有趣的是报道称增加酶和ATP的亲和力。其并未做分子动力学研究。
本文采用Gromacs 4.5.6版本
介绍:癌细胞介导细胞信号不仅位于细胞循环。而且阻止细胞凋亡。细胞凋亡要求特别的调节蛋白例如扁在蛋白酶系统(ubiquitin-proteasome system UPS)作为监测点,UPS包括E3遍在蛋白酶联体称之为CUllin-RING Ligases(CRLs),其核心为NEDD8(ubiquitin-like protein ;ULP)。
NEDD8修饰能够介入细胞循环和细胞骨架的调节。
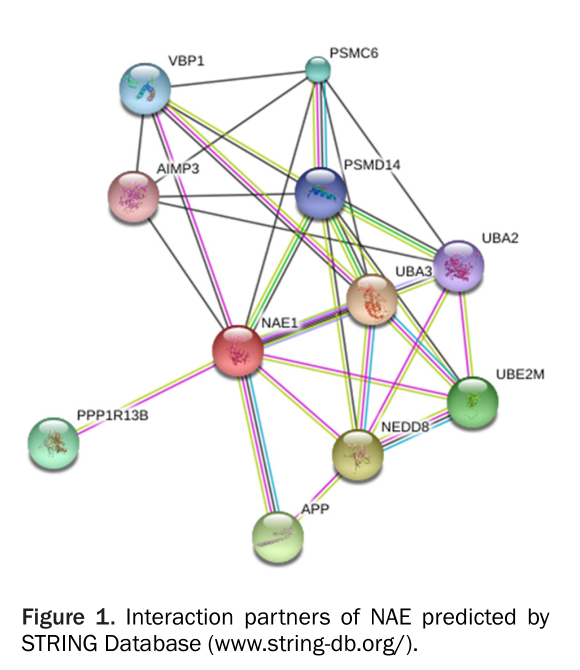
E1(NEDD8激活酶NAE)催化带有NEDD8的cullin的初始化。NAE与其他蛋白的联系阐述如图1.NAE的抑制剂MLN4924被揭露其阻止NAE的激活以及随后的cullins表达。被认为是一个潜在的抗癌药物,表达临床效用在一些固体肿瘤和血液病和近期发现淋巴瘤和多发性骨癌。
方法:
预测NAE可能的相互作用伙伴
使用STRING数据库(http://string-db.org/)。这个相互作用包括直接(物理)和间接(功能性)联系。
MLN4924属性计算和化学结构
通过Pfam database (www.http://pfam.xfam.org/),我们计算Molecular Weight,Molecular Formula, XLogP3, Hydrogen Bond Donor Count, Hydrogen Bond Acceptor Count,Rotatable Bond Count and etc。以及化学结构
分子动力学和分子结构最小化
使用之前教程方法,突变使用Modeller 9.13项目,mutant Y352H and I310N,MD 20ns,抑制剂和蛋白均为晶体结构
结果与讨论
蛋白联系预测
计算参数结果和化学构象
其说对于MD非常有用,个人觉得没什么用
分子动力学
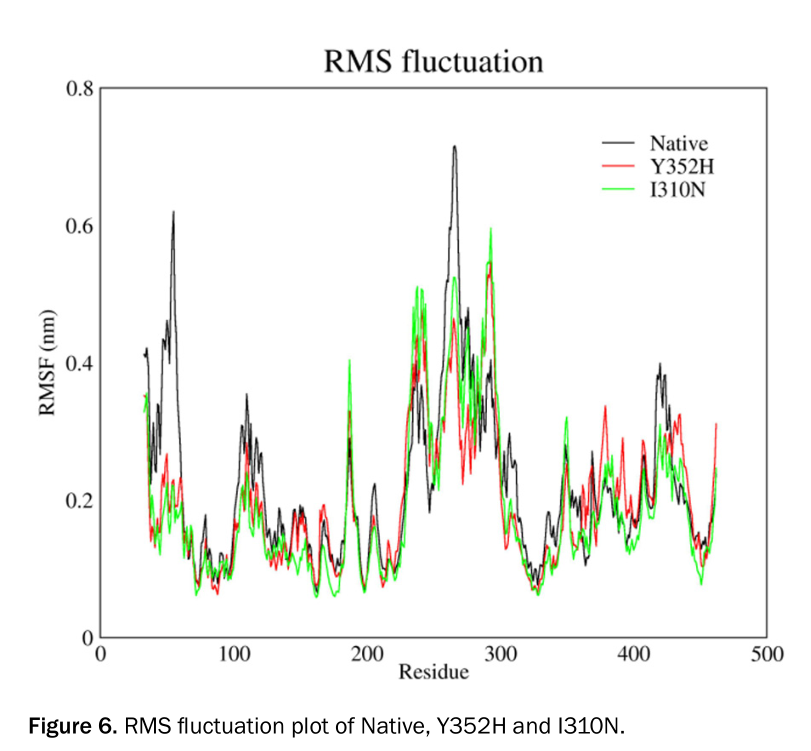
RMSD分析用来查看动力学稳定性(原文In MD analysis, Root Mean Square Deviation (RSMD) of simulated structure upon time is an appropriated and common criterion to verify Molecular dynamics stimulation stability.)
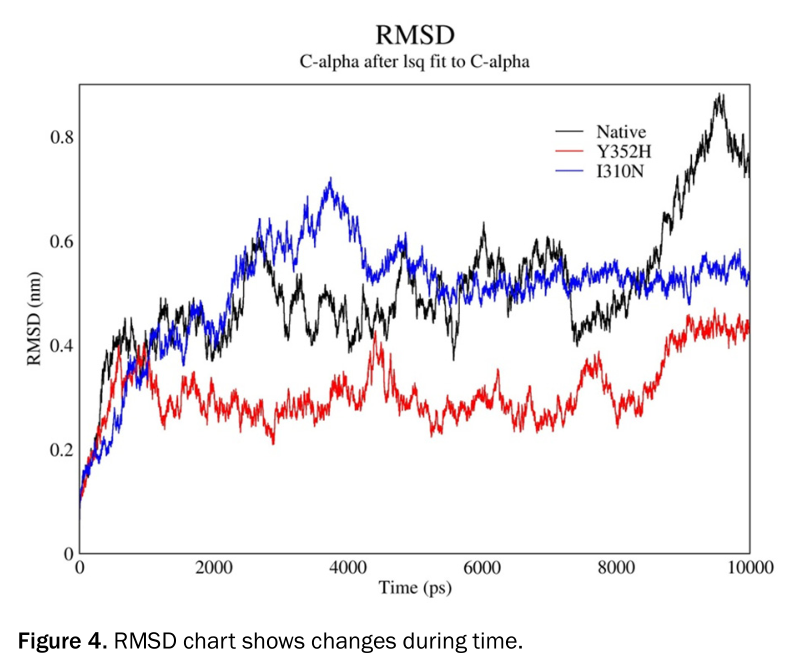
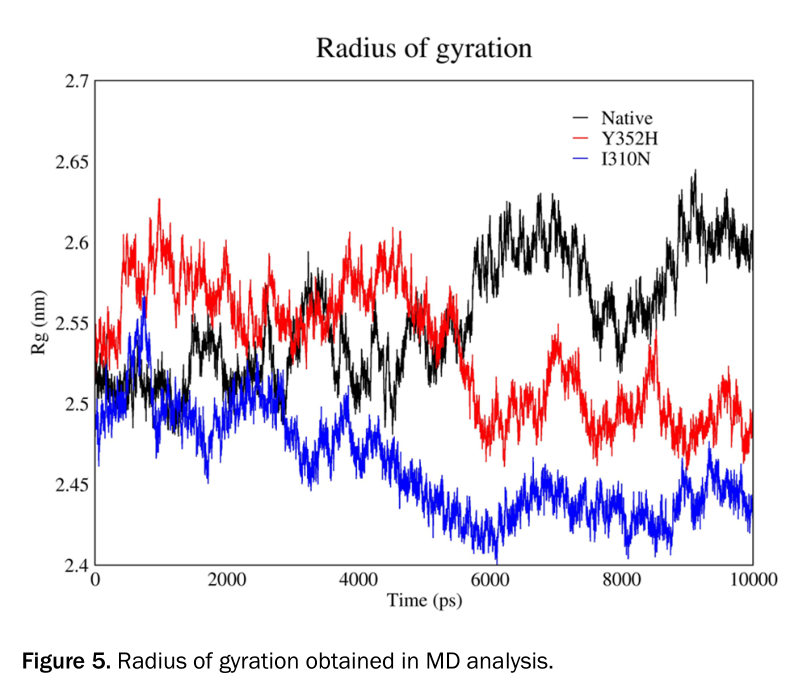
此外,蛋白结构域和形状可以被通过Gyration Radius计算修饰。这个参数提供了关于蛋白总体积分配,换而言之,其表明了紧密的分子形状?(tensed molecular shape)
RMSF 一些结构的柔性改变和改变的模式对于正常的与突变的不同的模式能够从此图中比较出来