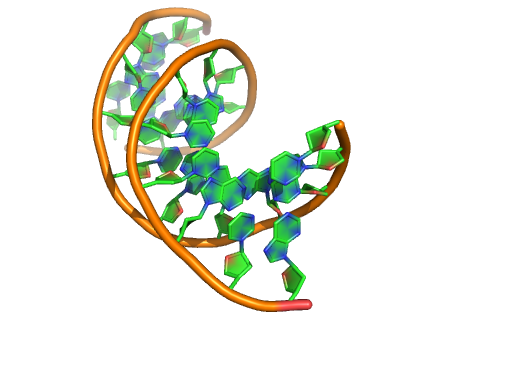
大家都知道chimera的核酸绘图功能非常强大,一般大家理解的pymol核酸绘图功能就显得薄弱了,但是今天在看pymol的cartoon的时候无意间看到了其核酸绘图,发现实际上其功能还是不错的。
默认设置
默认的一般有一个磷酸骨架和一个跨过核苷酸平面的枝棒,默认设置如下:
我们以PDB265d为例
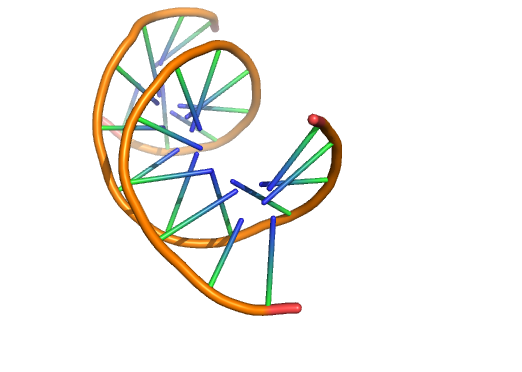
Cartoon 环模式(ring mode)
设置
set cartoon_ring_mode, value
值影响
0 枝条从骨架原子到嘌呤的N1或嘧啶的N3
1 简单的核糖平面和覆盖环键之间区域的基环
2 简单的核糖平面和覆盖环键内侧之间区域的基环(和模式1相比略小)
3 用棍棒包围核糖和基环的平面
4 在核糖中心和每个基环上的大直径环球体
5 大约为4的1/10的球体
例子
模式1: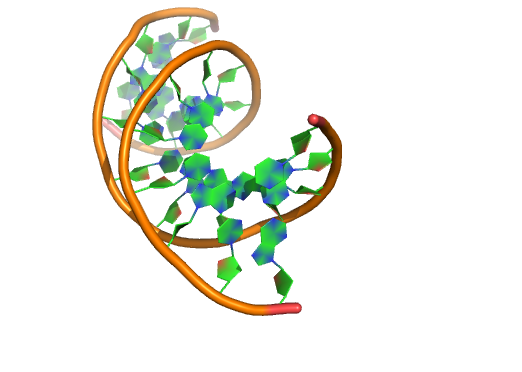
模式3: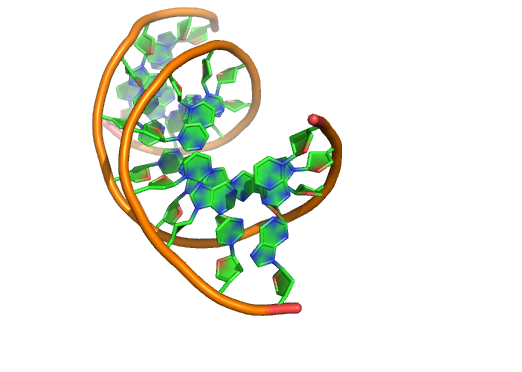
模式4: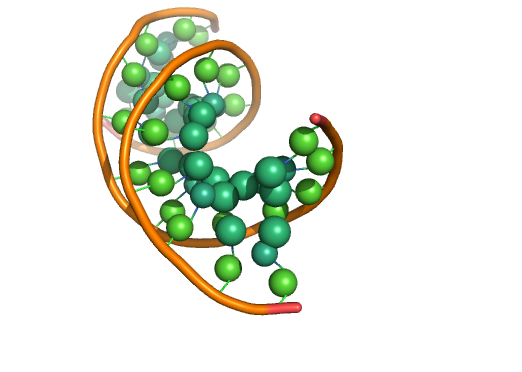
Cartoon ring finder
设置
set cartoon_ring_finder, value
值影响
0 没有环或枝条加入
1 核糖和基础环
2 只有基础环
3 和模式1类似,轻微的透视
4 和模式1类似,核糖核苷酸和碱基,以及蛋白质的芳香侧链显示
5 只有枝条,无环
例子
模式0:
模式1: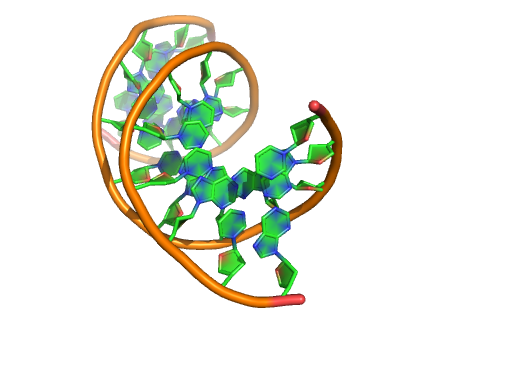
模式2: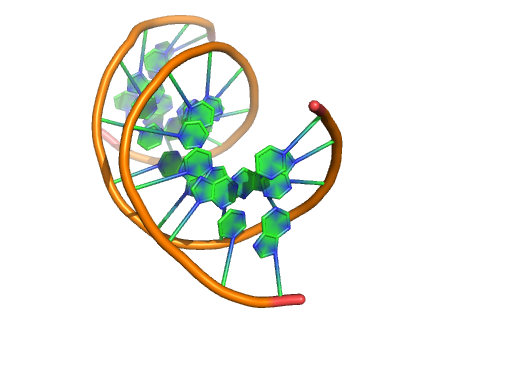
Cartoon梯子模式
设置
set cartoon_ladder_mode, value
值影响
0 没有枝条展示
1 枝条展示
例子
模式0: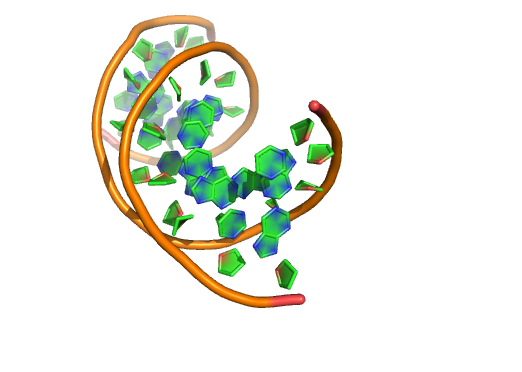
模式1:
Cartoon核酸模式
设置
set cartoon_nucleic_acid_mode, value
值影响
0 光滑的骨架通过磷原子,骨架的两端均终止于磷末端
1 光滑的骨架通过核糖C3’原子,骨架的两端最后C3’终止
2 光滑的骨架通过磷原子,主链在5’末端终止于磷,在3’末端终止于O3’
3 和0模式相似?
4 和模式2相似?
例子
模式0:
模式1: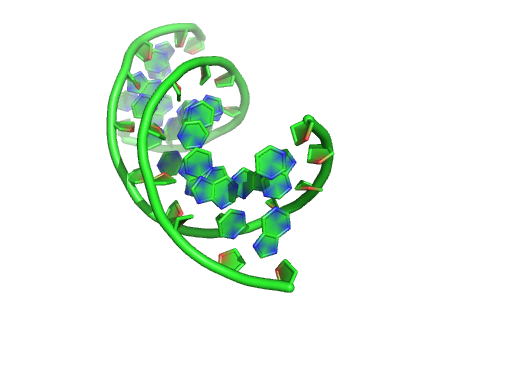
模式2: